Nat Commun:胡泽平/韩鹏/田艳涛团队合作构建基于代谢组学的胃癌诊断和预后机器学习模型
2024-04-09 测序中国 测序中国 发表于上海
研究团队揭示了胃癌患者血浆的代谢重编程图谱,发现基于代谢组学构建的机器学习模型能准确诊断胃癌患者,并预测患者预后风险。
胃癌是全球范围内的一种高度致命性癌症,及早诊断有助于改善患者的临床结果。内窥镜检查是临床诊断胃癌的金标准,但由于具有侵入性、成本高昂等不足,其临床应用在很大程度上受到了限制。同时,目前临床预后预测主要依赖于外科医生基于各种临床指标的经验判断,包括肿瘤位置、肿瘤分期信息和组织病理学,但这些指标的准确性有限。因此,我们迫切需要开发具有高灵敏度和特异性的非侵入性检测方法来检测胃癌,并更精确的预测患者结果,将其分层为不同的风险组以进行适当的干预。
近日,清华大学药学院胡泽平研究员、哈尔滨医科大学附属肿瘤医院韩鹏主任、中国医学科学院肿瘤医院田艳涛主任作为共同通讯作者,在Nature Communications发表文章“Metabolomic machine learning predictor for diagnosis and prognosis of gastric cancer”。研究团队揭示了胃癌患者血浆的代谢重编程图谱,发现基于代谢组学构建的机器学习模型能准确诊断胃癌患者,并预测患者预后风险。
研究团队建立了基于10个代谢物的胃癌诊断模型(10-DM)和基于28个代谢物的胃癌患者预后预测模型(28-PM)。验证分析显示,10-DM诊断模型即使对早期胃癌患者(stage IA)也能准确诊断,表现出比传统癌症蛋白标志物CEA,CA19-9,CA72-4等更优越的诊断效果(灵敏度0.925:0.428);28-PM预后模型表现出优于利用临床参数的传统模型的性能,并有效地将患者分层为不同的风险组,以指导精准干预。

胃癌的遗传和环境风险因素都会导致代谢变化,并进一步促进肿瘤的发生和发展。作为一种系统分析方法,代谢组学提供了代谢状态的综合概况,反映了遗传与环境相互作用的综合结果。机器学习作为一种人工智能方法,在许多生物医学领域都有着广泛应用,其在解释组学数据、开发预测模型、识别生物标志物以及对患者进行精准医学分层方面有着特殊的优势。但在分析胃癌代谢组学数据并开发潜在生物标志物方面,机器学习的利用尚未得到充分探索。
01 患者数据收集和研究设计
该研究共收集了702名个体的血浆样本(图1),包括389名胃癌患者和313名非胃癌对照。研究团队使用基于液相色谱-质谱联用的靶向液相代谢组学方法获得了血浆样本的代谢组学谱图,总共检测到147种代谢物,包括氨基酸、有机酸、核苷酸、核苷等。
随后,研究团队比较了队列1中胃癌和非胃癌个体的代谢景观,并使用机器学习算法分析了代谢标志物与临床表型之间的关联。研究团队开发了一个名为10-DM的胃癌诊断模型,评估了该模型在区分胃癌患者和非胃癌对照方面的表现,并利用外部测试集2验证了该模型的稳健性。除了诊断模型外,研究团队还利用181名胃癌患者的代谢组学数据进行机器学习分析,进一步构建了一个预后模型(28-PM模型),将该模型与利用临床指标的传统方法进行对比,并评估了模型的风险分层能力。
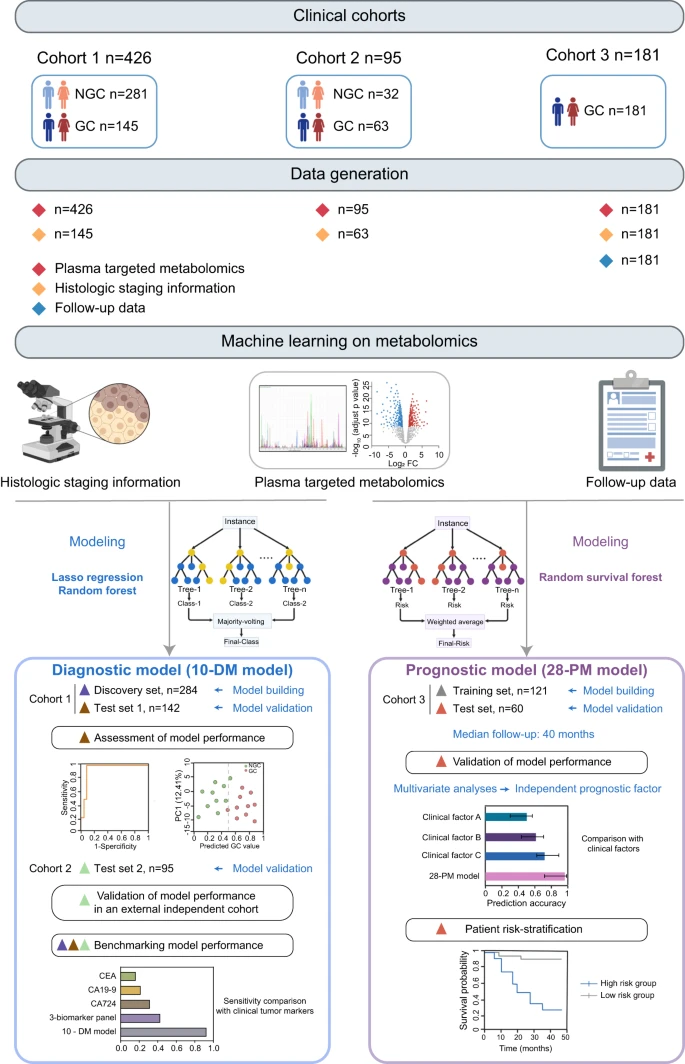
图1:研究概述。
02 胃癌患者血浆代谢分析
为了表征胃癌患者血浆代谢重编程的特征,研究团队对比了胃癌患者和非胃癌对照的代谢组学分析。结果显示,主成分分析(PCA)区分了胃癌样本和非胃癌对照样本,表明胃癌代谢组经历了重构(图2a)。共有45种代谢物在胃癌与非胃癌对照之间存在显著差异(图2b)。
有趣的是,随着疾病的进展,这些失调的代谢物表现出3种明显不同的趋势(Cluster 1-3)(图2c)。其中,Cluster 1中的代谢物(例如新葡萄糖甙和N(7)-甲基鸟苷)表现出持续增加,Cluster 2中的代谢物(例如谷胱甘肽二硫酸、尿苷和乳酸)显示持续下降。进一步分析发现了一系列严重扰乱胃癌患者的代谢途径,包括谷胱甘肽代谢、半胱氨酸和甲硫氨酸代谢(图2d),揭示了血浆代谢物在胃癌检测和预测中的潜在应用。
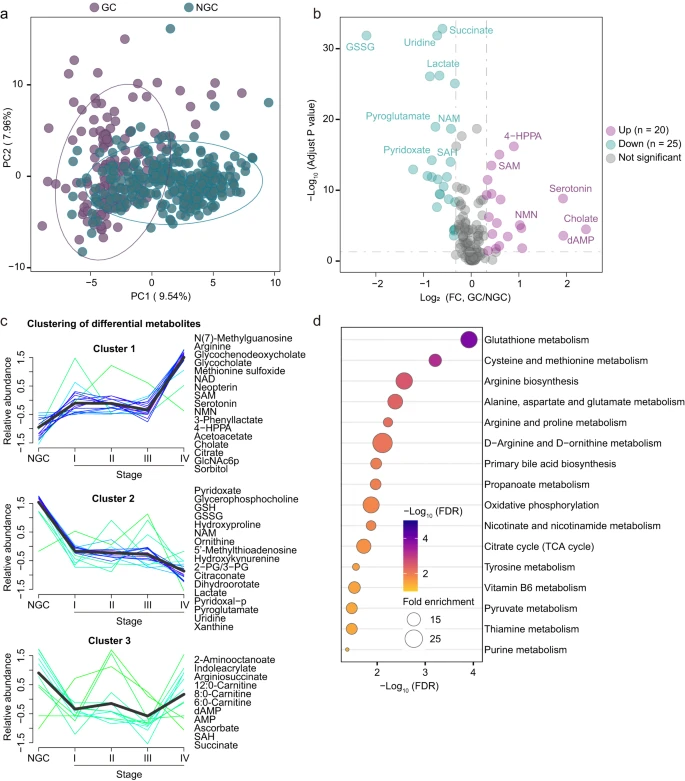
图2:与非胃癌对照相比,胃癌患者血浆代谢景观的重编程。
03 基于机器学习的生物标志物组可诊断早期胃癌
接下来,研究团队利用获取的重编程代谢谱来开发了创新的癌症诊断方法。研究团队选择了10种关键代谢物来区分胃癌和非胃癌对照(图3a),并利用这10个关键特征训练了一个随机森林模型,即胃癌诊断模型10-DM。经测试集1验证,获得到的ROC曲线下面积(AUROC)为0.967(灵敏度0.854,特异性0.926)(图3b)。每种代谢物对10-DM模型的贡献相对均匀,其中琥珀酸、尿苷和乳酸是贡献最重要的三个代谢物(图3c)。肿瘤发生和发展过程中的相对丰度分析表明,所有10种代谢物在胃癌和非胃癌对照之间都有显著差异。
为了直观地展示模型的性能,研究团队将每个参与者的预测值与其实际疾病状态进行了比较,发现10-DM模型准确识别了85.4%的胃癌患者(测试集1)和90.3%的GC患者(测试集2)(图3d,e)。
研究团队还在测试集1中应用10-DM模型来区分IA/IB期胃癌和非胃癌对照。分析显示,10-DM模型对IA期患者的预测准确率为90.9%(灵敏度0.813,特异性0.926),对IB期患者的预测准确率为92.7%(灵敏度:1,特异性:0.926),表明该模型在筛查早期胃癌患者方面具有较强的鉴别能力(图3f)。在测试集2中,10-DM模型对早期(I期和II期)患者的检测准确率为83.6%(灵敏度0.931,特异性0.75)(图3g),对IA期患者的检测准确率为79.1%(灵敏度0.909,特异性0.75),具有较高的灵敏度和可靠性。
此外,研究团队将10-DM模型的预测准确性与3种现有临床肿瘤生物标志物CA19-9、CA72-4和CEA的预测准确性进行了基准测试。与10-DM模型的0.925相比,CA19-9、CA72-4和CEA的区分灵敏度分别为0.217、0.317和0.165。同时,研究人员使用不同的机器学习算法对10-DM模型的性能进行了基准测试,发现10-DM模型始终表现出最佳的模型性能。以上结果表明,10-DM模型比临床实践中常规使用的3-生物标志物组和代谢分析中用于检测胃癌患者的其他算法具有更高的准确性。
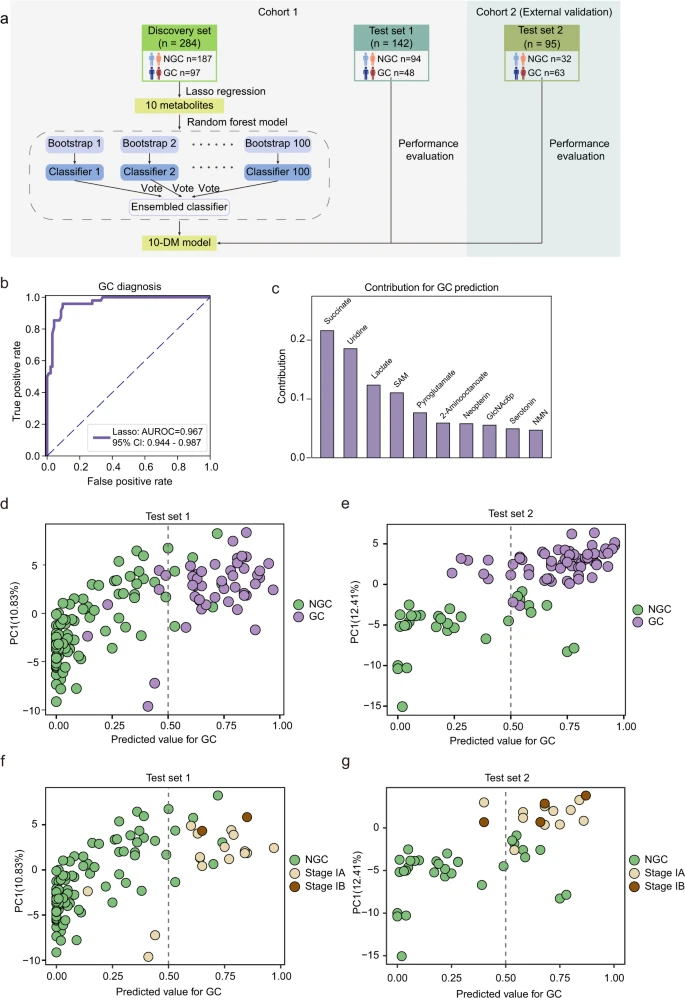
图3:用于胃癌诊断的10-DM模型。
04 代谢预后模型可以准确预测胃癌患者的预后
研究团队收集了来自181名胃癌患者的血浆代谢组学资料,利用随机生存森林方法建立了一个包含28种代谢物的预后模型(28-PM模型)(图4a)。在测试集上评估28-PM模型,发现其具有有效的预测能力,获得了0.832的AUROC(图4b)。有趣的是,研究发现这28种代谢物中有11种的相对丰度可以显著区分测试组患者的总生存期。总之,该机器学习衍生的预后模型在预测胃癌患者的临床预后方面表现出良好的性能。
此外,研究团队评估了28-PM模型与临床医生用于经验预后评估的三个临床因素相比的预测能力(图4c),并进一步尝试将临床特征的组合纳入28-PM模型,以评估这是否会增强28-PM模型的预测能力。结果显示,28-PM模型的预测效果均高于临床因素中的每一个(无论是单独考虑还是组合考虑),28-PM模型在预测胃癌患者不同阶段预后方面具有更强的稳健性(图4d);将临床特征纳入28-PM模型并没有对其性能产生实质性的改善。
研究团队进一步评估了28-PM模型对测试集中每个患者的预测性能,将胃癌患者分为高风险组和低风险组(图4e)。28-PM模型预测结果显示,高危患者的无病生存期和总生存期较低危患者差(图4f),高风险组的死亡个体比例更高,低风险组的非转移/非复发患者更突出(图4g),表明28-PM模型成功地识别了需要精细治疗方案的患者。
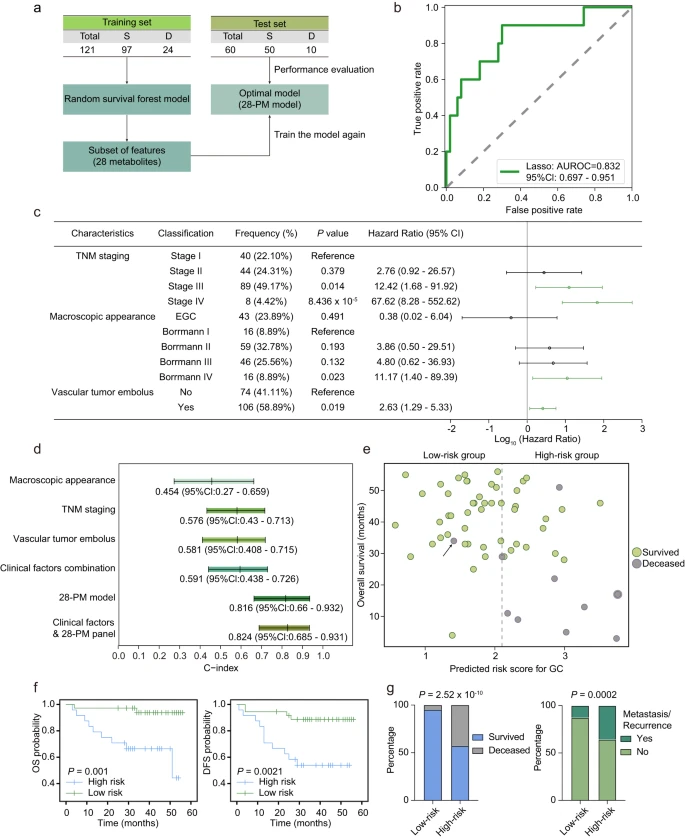
图4:预后模型在预测胃癌患者结果方面优于临床参数。
研究团队采用靶向代谢组学分析了来自多个中心的胃癌患者和非胃癌对照的血浆样本。利用代谢组学数据开发了一个机器学习诊断模型10-DM,该模型在识别IA期及其他期胃癌患者方面优于目前临床使用的癌症蛋白标志物。同时,研究团队还开发了预后模型28-PM,该模型显示出比采用临床指标的传统方法显著更高的一致性指数,表明其在预测临床结果方面表现更佳。
总的来说,该研究的发现揭示了胃癌的代谢景观,展示了应用机器学习分析代谢组学数据在胃癌早期检测和精准医学方面的优势。
论文原文:
Chen, Y., Wang, B., Zhao, Y.et al. Metabolomic machine learning predictor for diagnosis and prognosis of gastric cancer. Nat Commun 15, 1657 (2024). https://doi.org/10.1038/s41467-024-46043-y
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言














#胃癌# #代谢组学# #机器学习模型#
99