PNAS:谢晓亮课题组开发新型单细胞甲基化与羟甲基化测序技术
2023-12-19 测序中国 测序中国 发表于上海
该研究利用该技术描绘了小鼠早期胚胎发育过程中甲基化、羟甲基化以及半甲基化修饰的动态变化,揭示了小鼠早期胚胎甲基化重编程过程中主动/被动去甲基化的活力变化以及参与父母本基因表达调控机制的差异。
DNA甲基化的建立与去除通常关系到基因表达水平的变化,也会参与胚胎发育、疾病发生等过程。伴随二代测序技术进步,基于重亚硫酸盐(bisulfite)转化的全基因组DNA甲基化测序技术出现并且成为研究者观测基因组范围内DNA甲基化修饰分布的重要工具。陆续出现的单细胞DNA甲基化测序技术也多数基于重亚硫酸盐转化反应。但重亚硫酸盐会造成基因组DNA的严重片段化甚至降解,受此影响,基于重亚硫酸盐转化反应的单细胞DNA甲基化测序技术有低基因组覆盖率的缺点,同时由于重亚硫酸盐转化不能对甲基化(5mC)与羟甲基化(5hmC)修饰进行区分,在多能性细胞群(早期胚胎、胚胎干细胞)和神经细胞等羟甲基化修饰含量丰富的细胞类型中难以准确表征DNA甲基化修饰状态。因此,开发高基因组覆盖度的单细胞甲基化与羟甲基化测序技术是非常必要的。
近日,北京大学生物医学前沿创新中心(BIOPIC)/昌平实验室谢晓亮课题组在PNAS在线发表了题为“Single-cell bisulfite-free 5mC and 5hmC sequencing with high sensitivity and scalability”的研究论文,介绍了不依赖重亚硫酸盐转化的高基因组覆盖度单细胞甲基化与羟甲基化测序技术——Cabernet。该研究利用该技术描绘了小鼠早期胚胎发育过程中甲基化、羟甲基化以及半甲基化修饰的动态变化,揭示了小鼠早期胚胎甲基化重编程过程中主动/被动去甲基化的活力变化以及参与父母本基因表达调控机制的差异。

文章发表在PNAS
该论文所介绍的Cabernet技术中,研究团队整合Tn5转座、EM-seq酶学转化模块(NEB)实现单细胞DNA非甲基化胞嘧啶的转化,依赖转座序列为引物实现文库扩增及二代测序。通过和已发表技术进行对比,Cabernet甲基化检测准确性得以验证,同时还表现出了数据比对率高、基因组覆盖度高等方面的优势。此外,该论文一同介绍了Cabernet-H技术,实现了单细胞高基因组覆盖度的DNA羟甲基化测序。除了检测甲基化及羟甲基化修饰,该技术中Tn5转座反应对单细胞DNA进行片段化时,会在父本和母本基因组DNA上产生不同的断点,于是研究团队通过该断点信息实现同源染色体的区分,并进而实现在单条DNA分子中对于半甲基化修饰的检测。
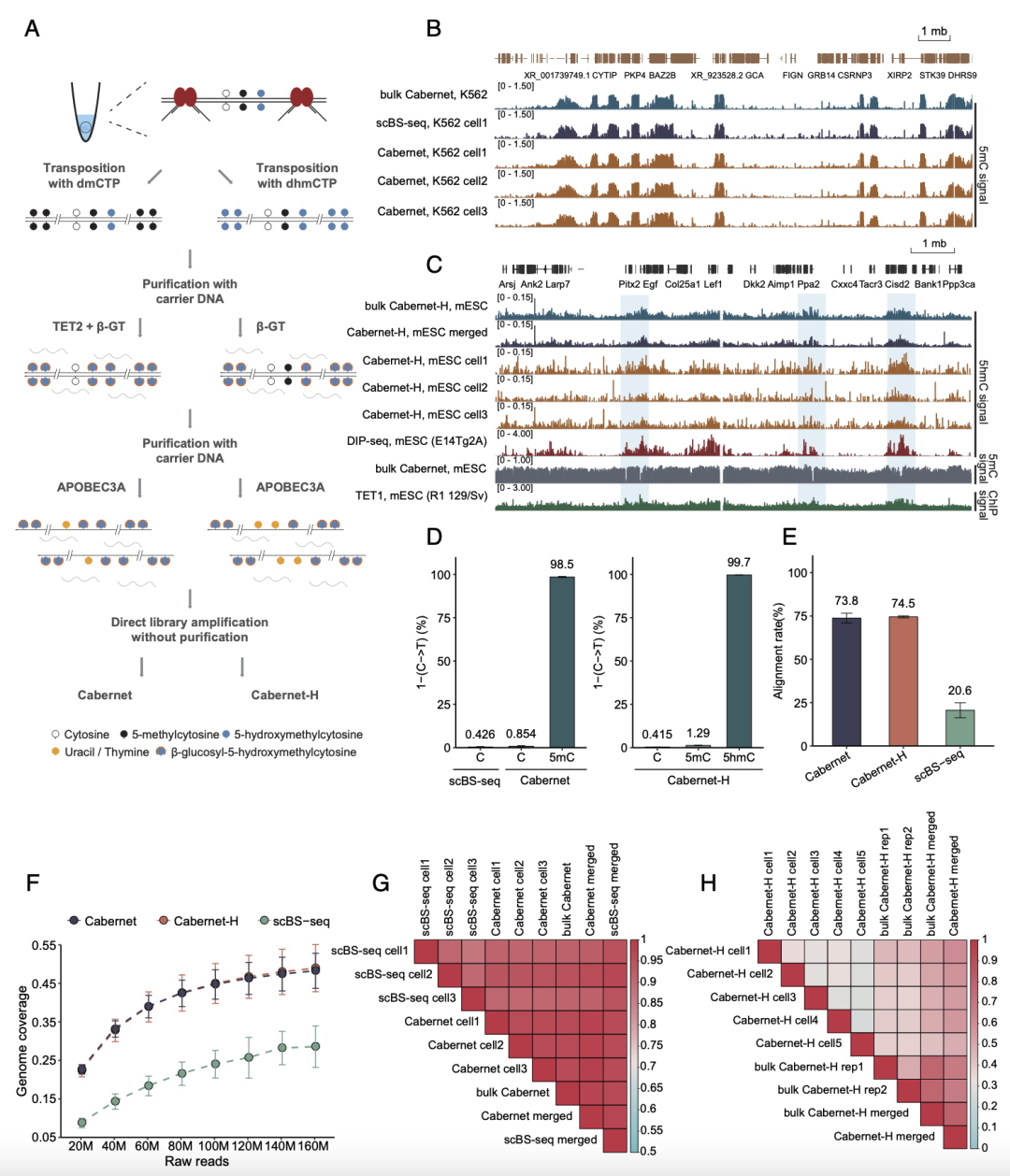
图1. 新型单细胞甲基化与羟甲基化测序技术原理及表现
研究团队利用该技术在小鼠早期胚胎发育过程中描绘了甲基化、羟甲基化以及半甲基化修饰的动态变化,解析了甲基化重编程过程中TET介导的主动去甲基化活力(羟甲基化修饰水平)以及复制依赖性被动去甲基化活力(半甲基化修饰水平)的变化。此外,该研究还揭示了主动/被动去甲基化参与父母本基因表达调控机制的差异。
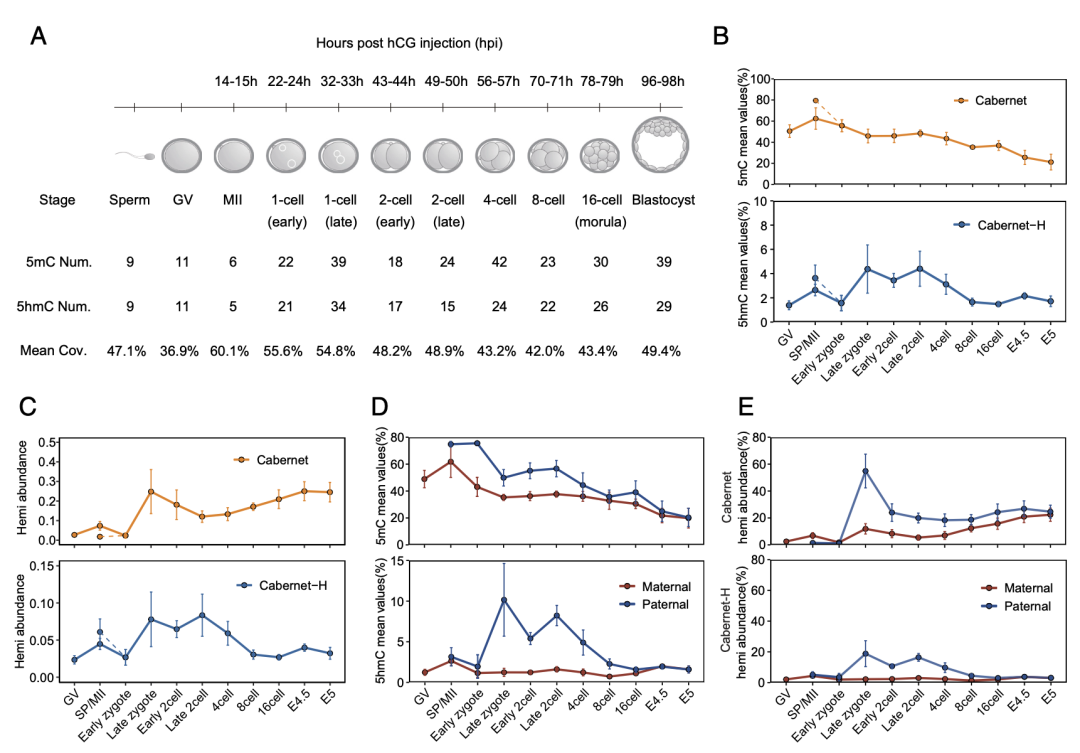
图2. 小鼠早期胚胎发育过程中甲基化、羟甲基化以及半甲基化修饰的动态变化
研究团队利用Tn5转座时引入序列标签组合,通过差异标签化的单细胞混合转化建库的方式进一步建立了高通量DNA甲基化检测平台。该方法兼备成本效益的同时,能够在两天时间高效实现数以千计的单细胞DNA甲基化检测。
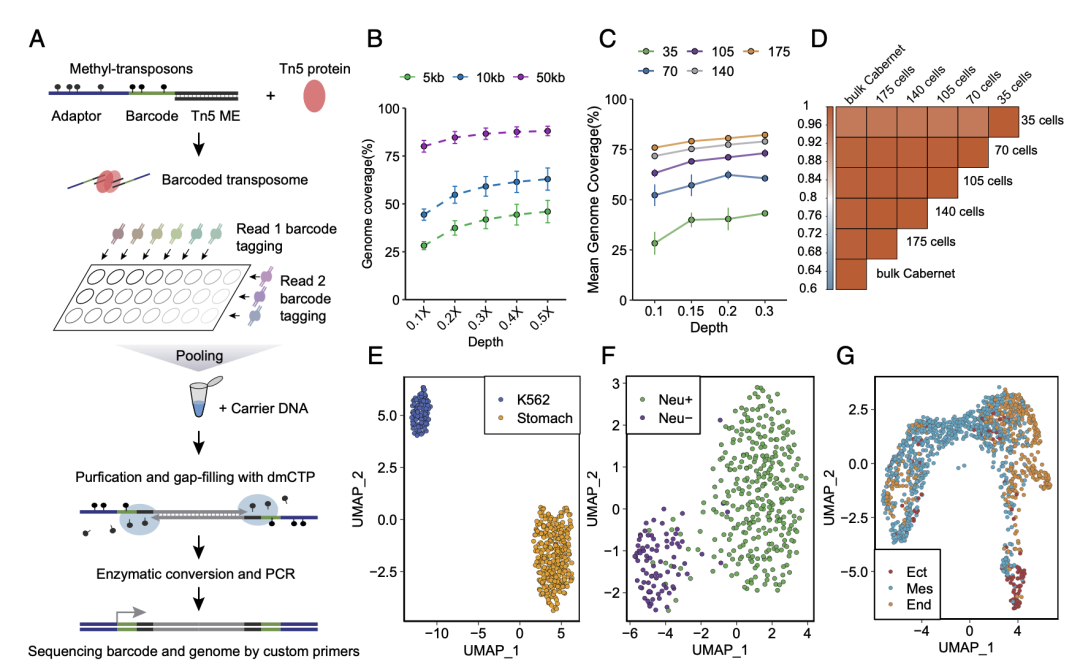
图3. 基于组合标签的高通量单细胞DNA甲基化检测平台
北京大学/昌平实验室谢晓亮教授为文章通讯作者;北京大学/昌平实验室研究员曹云龙、昌平实验室博士后白雅丽、北京大学博士生袁天骄为文章的共同第一作者。该研究得到了北京未来基因诊断高精尖创新中心和昌平实验室的资助。
论文链接:
https://doi.org/10.1073/pnas.2310367120
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#DNA甲基化# #单细胞甲基化# #羟甲基化测序技术#
91