深度:远见还是痴念?三代测序和它的时代丨奇点猛科技
2017-03-29 BioTalker 奇点原创文章(公众号:geekhe
自从本世纪初人类基因组计划完成之后,伴随着基因检测技术的飞速发展,在23andMe、ancestry和华大基因等公司,以及安吉丽娜·朱莉等明星的不断教育之下,很多人开始知道,为什么有些人喝酒脸红,为什么有些人更容易患乳腺癌。这些都与体内某些特定基因特定位点发生变异有关。现在依托于第二代高通量测序平台,我们已经开始尝试从基因的层面管理我们的生老病死。
本文为奇点原创文章(公众号:geekheal_com)
自从本世纪初人类基因组计划完成之后,伴随着基因检测技术的飞速发展,在23andMe、ancestry和华大基因等公司,以及安吉丽娜·朱莉等明星的不断教育之下,很多人开始知道,为什么有些人喝酒脸红,为什么有些人更容易患乳腺癌。这些都与体内某些特定基因特定位点发生变异有关。现在依托于第二代高通量测序平台,我们已经开始尝试从基因的层面管理我们的生老病死。
这一切都在二代测序的加持下,如火如荼的进行着。可总有一些「小意外」在不断的提醒我们:这还不够。
二代测序的软肋
几个月前(2016年12月2日),刊登在生物学预印网站BioRxiv上的一篇文章,在基因测序圈激起了不小的风浪。一名求医十余年,利用Illumina二代测序平台也没有找到病因的患者,被斯坦福大学Euan Ashley博士利用Pacific Biosciences(后文简称PacBio)的三代测序平台Sequel成功确诊为Carney综合征(1)。这是全球首次利用三代测序平台,通过全基因组测序在患者体内找到致病突变。标志着三代测序发展到了进入临床应用的拐点。
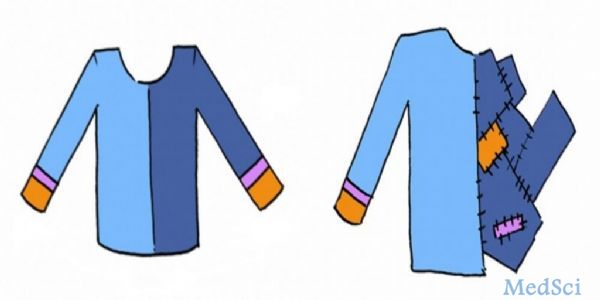
▲现存的所有测序技术都不具备通观人类基因组的能力。如上图所示,左图是衣服的标准品,右图一名裁缝学徒用同等布料制作的衣服。从整体上看,我们很容易发现学徒做的衣服在结构上与标准品有较大差异。然而,二代测序是个一次只能看长×宽为10cm×1cm的老师傅,于是他把学徒的衣服剪成10cm×1cm的布条,最后他可能发现,从学徒衣服上剪下来的布料,可以完好的拼装到标准衣服上。而三代测序则是一个一次能看长×宽为10cm×100cm的老师傅,当他采用同样的方法裁剪衣服在标准品上比对时,很容易就发现,学徒在右边衣服的设计上很有新意。由此观之,三代测序显然在检测结构变异上更具优势。
「二代测序对于SNP(single nucleotide polymorphism,单核苷酸多态性)和小于50bp的InDel(Insertion-Deletion,插入或缺失)变异检测相对比较准确,但是大的结构变异检测却非常困难。」国内首家三代测序服务公司武汉未来组生物科技有限公司创始人兼CEO汪德鹏告诉奇点网,「PacBio三代测序平台的读长是二代测序的100倍左右,可以非常好的解决基因结构变异问题。」
自从2005年二代测序技术实现商业化开始到现在,由于测序成本不断下降,研究人员已经在人体内发现了大量与疾病有关的基因突变,让我们对基因变异与人类健康和疾病之间的关系有了更深入的理解。然而,类似上面利用二代测序技术也不能给患者确诊的案例时有发生,这种现象不仅出现在遗传病的诊断上,还可能出现在寻找癌症发生的原因(2; 3),肿瘤耐药性产生的原因(4),以及精神分裂症(5; 6)等发生原因的探寻上。虽然早在上个世纪50年代中期就已经有研究人员利用核型分析(Karyotype analysis)观察染色体变异,但是经过近半个世纪的发展,人类对DNA的结构变异仍旧知之甚少。
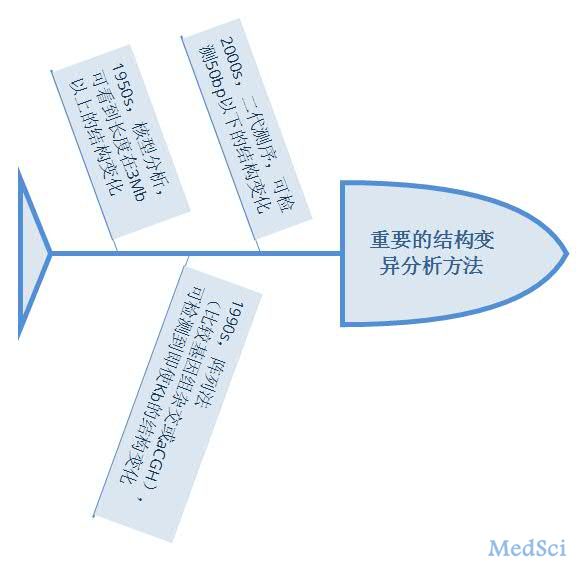
▲1950年以来DNA结构变异研究的方法,以及适用的检测范围,50bp-100kb这个范围的结构变异缺少很好的检测手段。
贝勒医学院的分子遗传学家Jim Lupski告诉《自然》杂志的记者Monya Baker的一段趣闻便是明证。早在1991年Lupski团队发现腓骨肌萎缩症(Charcot-Marie-Tooth,CMT),是由于某个特定的基因多了一个拷贝(结构变异的一种)导致的时候(7),学界一片哗然,那些打小就接受经典孟德尔遗传学教育的专家们无论如何都不认可Lupski这一发现。学界的不认同,直接导致顶级期刊《自然》和《科学》拒绝刊登Lupski的研究论文,最后他不得不用四种独立的实验方法证明他是对的,这才被《细胞》接收(8)。直到2004年Michael Wigler博士团队和Charles Lee博士团队,分别在《科学》(9)和《自然遗传学》(10)发表结构变异的开创性研究论文。自此,关于结构变异的研究论文开始变多,基因组变异数据库中结构变异的条目数量也逐渐增多(8)。
「非单核苷酸变异占到了人类基因组所有变异的74%,」Craig Venter研究团队在2007年发现(11)。研究人员逐渐意识到,人类遗传多样性的故事用二代测序是讲不完的,因为结构变异也是疾病的重要驱动力。「长读长对解决结构变异会有帮助,」博德研究所的Steve McCarroll博士说,他在DNA结构变异对于精神疾病之间关系的领域颇有建树(6)。
更符合精准医学使命的三代测序
目前市面上已经实现商业化的长读长测序平台主要有Illumina的Tru-seq Synthetic Long-Read、Oxford Nanopore Technologies的纳米孔平台MinION,以及PacBio的单分子实时(SMRT)测序平台,其中发展的最为成熟稳定的是PacBio的单分子实时(SMRT)测序平台(12)。
在SMRT测序平台的诞生的65年前,康奈尔大学诺贝尔物理学奖得主Hans Albrecht Bethe(1906-2006)发现了一个现象:当微孔的直径小于光的波长时,光在穿过微孔时光强会急剧衰减(13)。没成想,几十年后同在康奈尔大学的Watt Webb教授和PacBio的创始人Jonas Korlach和Stephen Turner将这一发现变成了一项技术,零模波导(zero-mode waveguide,ZMW)技术(14)。经过几年的摸索,Webb教授团队发现,ZMW技术可以用来检测并观察溶液中的单分子。2003年《科学》杂志以封面的形式报道了这一研究成果(15)。
一年之后,Korlach和Turner组建PacBio公司。当时人类基因组计划结束时间并不久,人们从基因组计划中看到了生命科学的未来,但是一代测序超低的通量和超高的成本,让研究人员难以前行。寻找通量高、速度快、价格低的测序方法,成为很多研究人员的梦想。当此之时,罗氏、Illumina和ABI的二代测序技术还在紧锣密鼓的研发当中。PacBio也想在测序市场占有一席之地。经过6年的开发,PacBio基于ZMW技术的SMRT测序平台终于在2009年初诞生了,科学杂志再一次刊登了这一重要发明(16)。
▲2009年二代测序完成一个人的基因组需要10万美元左右
「在09年的时候,有一阵子PacBio的测序炒的很厉害,那个时候大家认为,以后会是15分钟测完一个人的基因组,成本1000美元,而且读长很长。当时的定义,之所以叫三代,是因为有几个特征,读长长,测序速度快,测序价格要便宜。」汪德鹏说。伴随着研究人员对SMRT测序平台的殷殷希望,PacBio在2010年10月上市了,股价接近17美元,累计募集2亿美元。半年之后,PacBio终于推出全球首套三代单分子实时测序系统PacBio RS,标志着三代测序进入新时代。当时,以Illumina为首的二代测序平台个人全基因组的测序价格刚刚跌破一万美元大关。随后,一些研究机构开始使用PacBio RS,北京的药用植物研究所也在RS发布半年之后,购买了亚洲第一台RS测序仪。然而,世界各地的研究人员很快发现,RS根本就没有PacBio承诺的那么美好,价格昂贵不说,错误率还高,读长也并没有比一代测序好多少。显然,PacBio过度承诺了。在2012年底,PacBio的股价跌至接近1美元。

▲PacBio股价走势图
尽管如此,还是有人从中看到了机会。「至今,还有很多人对SMRT测序平台的认识只停留在2009年:PacBio的测序平台错误率约15%,太高了,不能用。」汪德鹏说。汪德鹏在2011年组建了中国第一家提供三代测序服务的公司武汉未来组,四年之后汪德鹏和哥伦比亚大学生物信息学副教授王凯共同组建了北京希望组,希望组是全球首家将三代单分子实时测序技术应用于疾病检测的企业。去年年底,北京希望组完成由经纬中国和赛富投资领投的近亿元人民币A轮融资,将全力推动三代测序技术应用和普及。「机会就是当大部分人觉得不靠谱的时候,你却发现了它其实是靠谱的,」汪德鹏说。
三代测序的机会在哪里?
经过两年的改进升级,PacBio在2013年初推出了RS II,测序数据通量翻一番,其他方面的性能也有一定的改进。就在这时,在二代测序市场失利的Roche,意欲借助PacBio杀入人体基因检测体外诊断领域(IVD),与PacBio签订了高达7500万美元的战略合作协议,根据协议PacBio需要开发基因检测临床产品,Roche将获得产品的全球独家销售权。在制药巨头Roche的加持下,PacBio股价飞涨73%。
到2015年底PacBio与Roche共同推出Sequel测序平台,新的平台通量较上一代产品提升了7倍,另外机器成本也在大幅下降。罗氏测序部门的负责人Dan Zabrowski表示,这款测序平台将作为罗氏测序仪器的基础,最初用于临床研究,之后才用于体外诊断。然而,Sequel推出一年之后,Roche发现Sequel并不能满足他们进军体外诊断市场的野心,于是与PacBio终止了合作协议。受此影响,PacBio股价下跌超过43%。
「罗氏觉得PacBio发展太慢那是因为罗氏需要一个非常大的体量的这样一个市场。坦率的讲,三代测序的确没有达到罗氏希望看到的那个程度,毋庸置疑,现在的主流还是二代测序。但是三代测序对于我们这样一个公司而言,这个体量是非常适合的。」汪德鹏说,「另一个原因是,罗氏当时是打算利用PacBio做HLA和HPV、HCV、HIV病毒等领域的诊断,这个目前还不符合PacBio主要进攻的方向。」
▲汪德鹏
然而,西边不亮东边亮,就在Roche与PacBio分手之后,北京希望组宣布采购6台Sequel测序仪,诺禾致源宣布购买10台Sequel测序仪。「PacBio的测序平台在基因组领域的应用日趋成熟,我们参加国际会议时发现,现在已经有很多科学家在使用PacBio的测序平台,」诺禾致源副总裁吴俊告诉奇点网,「在购买Sequel之前,诺禾致源实际上也提供三代测序服务,但是我们的技术评估发现这个平台还不够稳定,所以没有购买。去年年底,升级后的Sequel平台更加稳定,数据量和读长也有显着的提升。我们估算了一下三代测序科研市场的2017年的增量,感觉今年采购的10台仪器很有希望满负荷运行。」毋庸置疑的是,三代测序现在的主要应用场景是还是科研,要走向临床还需要很多科学研究和技术积累,而这个领域国内外的研究机构和企业正在持续不断的推动。吴俊说,「和二代测序一样,三代测序在医学上的应用也需要一个技术研发和普及的过程。任何一项技术从科研服务转向临床应用都需要一定的时间。诺禾致源目前已经占据了科技服务一半的市场份额,我们希望新采购的Sequel平台在近一年内对科研有所推动。」2016年,武汉未来组参与完成的第一个亚洲人参考基因组「华夏一号」在线发表于《自然通讯》上(17)。汪德鹏介绍,「华夏一号」是用三代测序在做,是黄种人的一个参考序列,而且是非常精准的参考序列,它弥补了以前国际参考序列里面存在的200多个洞,此外,他们还找到了 2万多个结构变异。更重要的是,他们发现有一些未知基因序列有转录和表达,但是因为GC含量的关系在Illumina测序中完全看不到,这些全新的基因是以前所不知道的。这再次表明,人类基因组还有太多的信息被我们遗漏掉。这些信息与疾病有什么关系,我们还一无所知。「与二代测序相似,三代测序在临床医学的应用需要一个长期的过程,我们认为未来2-3年内三代测序会出现临床应用的成熟产品。另外,我觉得Sequel的出现对Pacbio意义重大,」经纬中国医疗组的孙凌皓说,「长读长的成本降低是大的趋势,我不觉得三代测序和二代测序是互相取代的关系,测序技术发展到现在,一代测序仍然有很好的应用场景。但一代测序和二代测序的局限也给三代测序留下了巨大的市场空间。」
北京希望组已经在跟一些具体应用领域的医生一起做三代测序的应用开发。例如,在HLA骨髓配型这个领域,他们在跟中国最大的白血病治疗医院道培医院集团合作,利用三代测序开展HLA分型。此外,作为世界首家三代测序遗传病检测公司,希望组还在跟中日友好医院的顾卫红大夫一起在SCA(脊髓小脑共济失调)领域进行科研合作。
前面Ashley团队的研究成果表明,长读长测序在诊断遗传病上确实具备二代测序无可比拟的潜力。过去几年的研究表明,二代测序做遗传病有很大的局限,检出率在30%-50%左右。「我们希望利用三代测序来改变这个状况,如果我们能够把检出率提高20%-30%,那意义就非常大了。当然,这也是我们的梦想,还没有变成现实。」汪德鹏说。

▲Euan Ashley
目前看来,制约三代测序技术大范围应用的主要因素还是成本。「三代测序现在做医学检测价格还比较高,没有那么容易。」吴俊说。据悉,目前三代测序的成本是二代测序数十倍。然而,这对于动辄需要3-5年时间才能诊断的罕见病而言,哪一种方法带来的好处多,似乎是显而易见的。不过,根据事物的基本发展规律,三代测序的价格下降是必然的。「如果成本够低的话,大家还是偏向于可以获取信息更多的方法,」吴俊说。在接受Mendelspod采访时,华盛顿大学Evan Eichler教授更有惊人的言论,「如果三代测序的成本降到二代的两倍,二代测序的死期就到了。」尽管现在大部分人都认为三代测序不会取代二代测序,但是我还是认为,能够提供更多基因组信息的三代测序,甚至四代测序肯定是未来。到时,人人都会有一个信息无比丰满的个人基因组数据库。
原始出处:
【1】Merker J, Wenger AM, Sneddon T, Grove M, Waggott D, et al. 2016. Long-read whole genome sequencing identifies causal structural variation in a Mendelian disease. bioRxiv
【2】Yao F, Kausalya Jaya P, Sia Yee Y, Teo Audrey SM, Lee Wah H, et al. 2015. Recurrent Fusion Genes in Gastric Cancer: CLDN18-ARHGAP26 Induces Loss of Epithelial Integrity. Cell Reports 12:272-85
【3】Zhang X, Choi PS, Francis JM, Imielinski M, Watanabe H, et al. 2016. Identification of focally amplified lineage-specific super-enhancers in human epithelial cancers. Nat Genet 48:176-82
【4】Smith CC, Wang Q, Chin C-S, Salerno S, Damon LE, et al. 2012. Validation of ITD mutations in FLT3 as a therapeutic target in human acute myeloid leukaemia. Nature 485:260-3
【5】Schizophrenia Working Group of the Psychiatric Genomics C. 2014. Biological insights from 108 schizophrenia-associated genetic loci. Nature 511:421-7
【6】Sekar A, Bialas AR, de Rivera H, Davis A, Hammond TR, et al. 2016. Schizophrenia risk from complex variation of complement component 4. Nature 530:177-83
【7】Lupski JR, de Oca-Luna RM, Slaugenhaupt S, Pentao L, Guzzetta V, et al. 1991. DNA duplication associated with Charcot-Marie-Tooth disease type 1A. Cell 66:219-32
【8】Baker M. 2012. Structural variation: the genome’s hidden architecture. Nat Meth 9:133-7
【9】Sebat J, Lakshmi B, Troge J, Alexander J, Young J, et al. 2004. Large-Scale Copy Number Polymorphism in the Human Genome. Science 305:525-8
【10】Iafrate AJ, Feuk L, Rivera MN, Listewnik ML, Donahoe PK, et al. 2004. Detection of large-scale variation in the human genome. Nat Genet 36:949-51
【11】Levy S, Sutton G, Ng PC, Feuk L, Halpern AL, et al. 2007. The Diploid Genome Sequence of an Individual Human. PLOS Biology 5:e254
【12】Lee H, Gurtowski J, Yoo S, Nattestad M, Marcus S, et al. 2016. Third-generation sequencing and the future of genomics. bioRxiv:048603
【13】Bethe H. 1944. Theory of diffraction by small holes. Physical Review 66:163
【14】Levene MJ, Korlach J, Turner SW, Craighead HG, Webb WW. 2007. Zero-mode waveguides. Google Patents
【15】Levene MJ, Korlach J, Turner SW, Foquet M, Craighead HG, Webb WW. 2003. Zero-Mode Waveguides for Single-Molecule Analysis at High Concentrations. Science 299:682-6
【16】Eid J, Fehr A, Gray J, Luong K, Lyle J, et al. 2009. Real-time DNA sequencing from single polymerase molecules. Science 323
【17】Shi L, Guo Y, Dong C, Huddleston J, Yang H, et al. 2016. Long-read sequencing and de novo assembly of a Chinese genome. Nature Communications 7:12065
版权声明:
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言