Nat Commun:性能优于传统生物标志物!基于网络生物学的机器学习方法可准确预测癌症患者免疫治疗反应
2022-08-13 测序中国 测序中国
结果显示,基于NetBio的ML表现出一致的优良预测性能,并且优于纯数据驱动方法识别的特征。
免疫疗法通过激活人体的免疫系统来对抗癌细胞,主要包括免疫检查点抑制剂(ICI)、过继性细胞转移疗法(ACT)、肿瘤特异性疫苗等。与传统抗癌药物相比,其副作用较小。此外,免疫疗法还可以利用免疫系统的记忆和适应性,使受益于该疗法的患者能够获得持续的抗癌效果。
近年来,ICI极大地改善了癌症患者的临床治疗、提高了患者的生存率。但目前只有少数患者(约占30%实体肿瘤患者)可以从中受益,绝大多数患者对ICI没有反应。已有的ICI反应相关生物标志物通常不能准确预测ICI治疗反应。
网络生物学是识别可靠生物标志物的一种强有力手段。具有相似表型作用的基因倾向于在蛋白质-蛋白质相互作用(PPI)网络的特定区域共定位,这一特性已被用于识别能预测表型结果的基因模块。基于网络的方法提供了高预测性和低噪声的生物标志物,但该方法预测大样本癌症患者对ICI治疗反应的有效性尚未得到验证。
近日,韩国浦项科技大学、延世大学的研究团队在Nature Communications发表了题为“Network-based machine learning approach to predict immunotherapy response in cancer patients”的文章。研究团队提出了一种基于网络生物学的机器学习(ML)模型,可以对ICI数据集进行稳健的预测,并识别潜在的网络生物标志物(NetBio)。研究团队整理了700多例接受ICI治疗的患者样本,包括临床结果和转录组数据。在基于NetBio的预测中,该方法准确预测了三种不同癌症(黑色素瘤、胃癌和膀胱癌)对ICI治疗的反应。此外,基于NetBio的预测效果优于基于其他传统生物标志物(如ICI靶标或肿瘤微环境相关标志物)的预测。

文章发表在Nature Communications
主要研究内容
基于网络的免疫治疗反应预测概述
研究团队选择ICI靶点附近的通路来识别与ICI反应相关的生物学通路,并使用STRING PPI网络进行了模型的构建。首先,研究团队使用ICI靶标作为种子基因,在生物学网络上呈递ICI靶标的影响,网络呈递的一个特点是,越接近ICI靶标的节点影响分数越高。接下来,选择影响分数高的前200个基因,并鉴定其富集的生物通路,通过使用所选择的生物通路来预测免疫治疗反应,并将这些通路定义为NetBio。
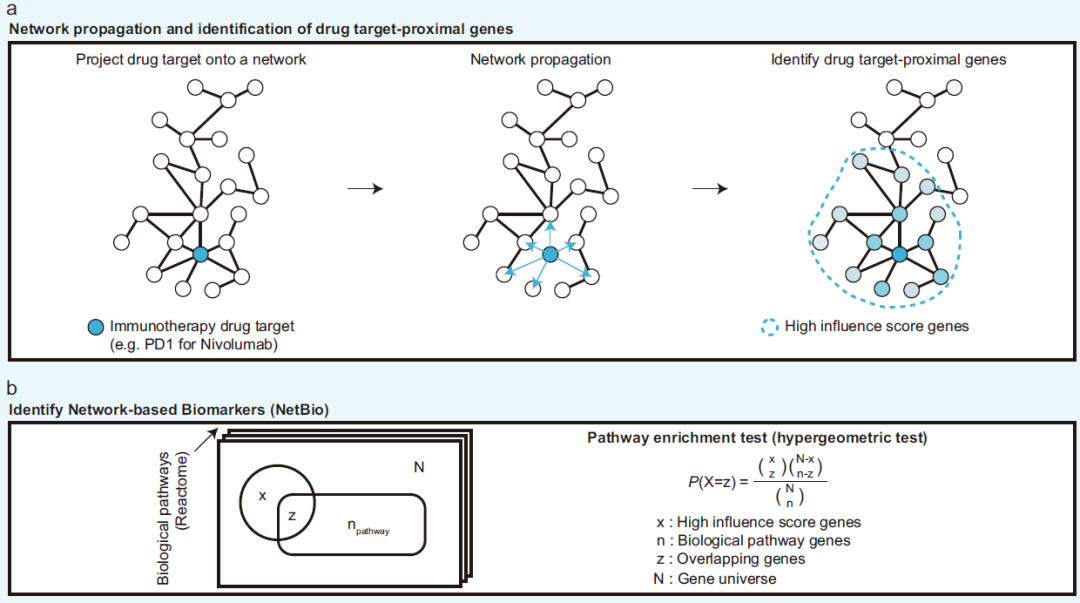
图1. 基于PPI的特征基因选择模式图。来源:Nature Communications
为了进行基于ML的免疫治疗反应预测,研究团队使用NetBio作为输入特征,并将基于基因的生物标志物(即免疫治疗靶基因)、基于肿瘤微环境的生物标志物或从数据驱动的ML方法中选择的通路作为阴性对照,通过监督学习训练ML模型(图2)。
研究团队使用不同的训练数据集和测试数据集的组合广泛地评估了预测性能的一致性。具体而言,1. 研究内预测,其中训练和测试数据集来自单个队列;2. 跨研究预测,其中两个独立的数据集被用作训练和测试数据集。此外,研究团队还交替使用大量或少量的训练样本来衡量不同训练条件下预测性能的一致性。
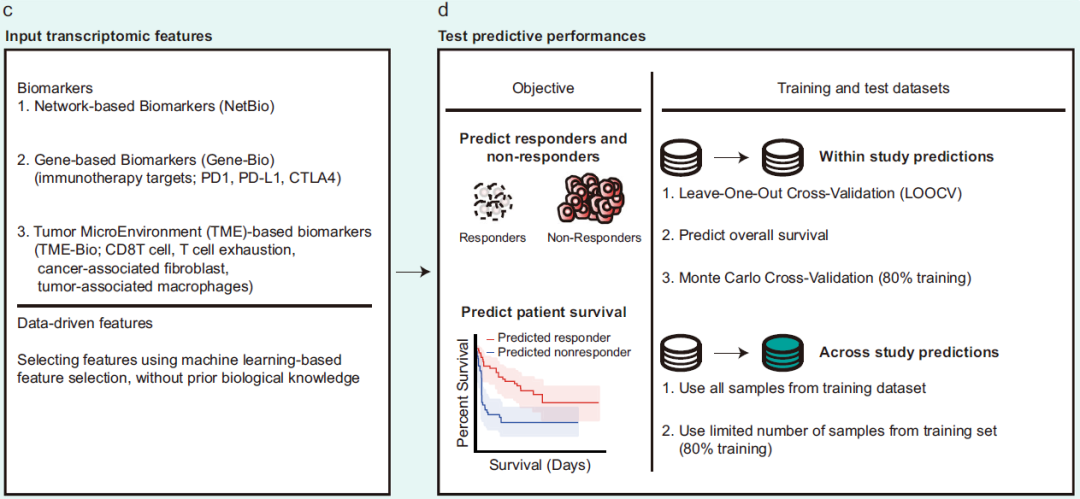
图2. 基于NetBio的机器学习模式图。来源:Nature Communications
基于NetBio的ML可以对ICI治疗反应和总生存期做出一致预测
研究发现,NetBio转录组预测ICI反应时的预测性能一致,基于药物靶标表达水平做出的预测不太一致(图3)。使用NetBio训练的ML模型在所有4个数据集(两个黑色素瘤队列、一个转移性胃癌队列和一个膀胱癌队列)中始终保持准确的预测;药物靶标仅能在黑色素瘤队列中实现准确预测。
在三个具有总生存数据的数据集中,基于NetBio的ML预测的ICI应答患者总生存时间显著延长。研究团队还比较了NetBio与其他已知的ICI相关生物标志物的预测性能,发现在大多数情况下,基于NetBio的预测效果优于其他方法。
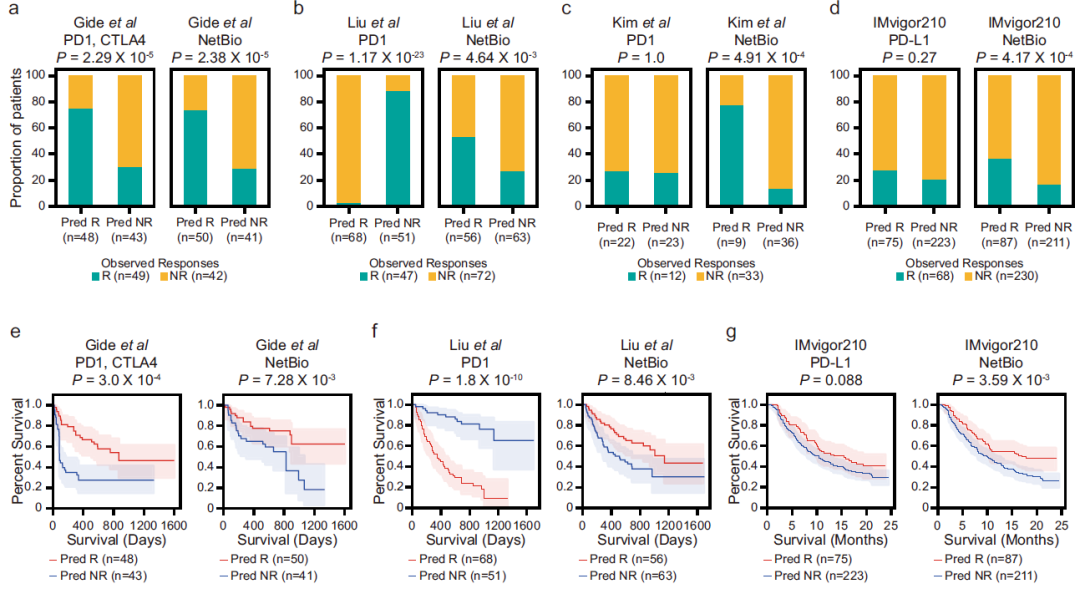
图3. NetBio在对ICI治疗反应和总生存期层面的预测。来源:Nature Communications
NetBio预测优于纯数据驱动的特征选择方法
研究团队测试了与纯数据驱动的特征选择方法相比,添加了代表PPI网络先验生物知识的NetBio能否改善特征选择。结果显示,与纯数据驱动的ML预测相比,基于NetBio的ML模型能够持续提高预测性能。具体而言,在11个不同的任务中,研究团队发现NetBio的预测性能明显优于基于纯数据ML的特征选择。此外,在对黑素瘤队列进行预测时,均观察到性能改善,表明网络引导选择有助于减少ML模型的过拟合。上述结果表明,利用基于网络的生物标志物选择能够识别可靠的转录组生物标志物。
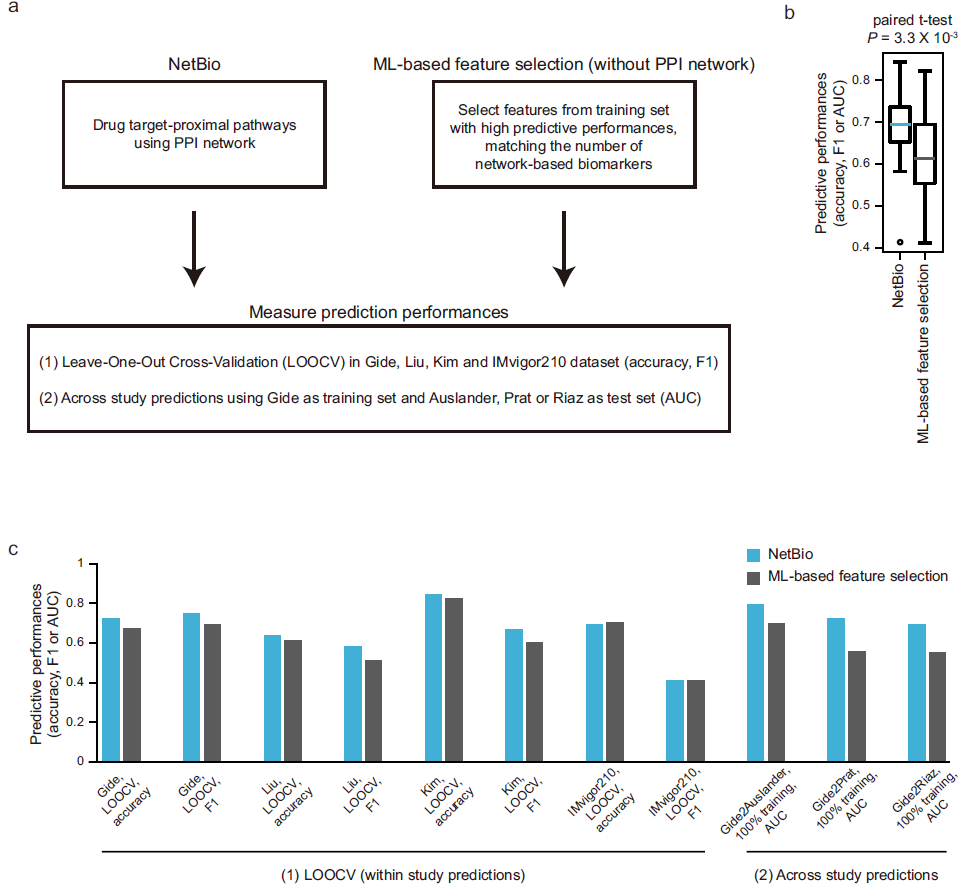
图4. NetBio与纯数据驱动模型的性能比较。来源:Nature Communications
NetBio预测再现了TCGA数据集中的免疫微环境
由于NetBio在包含三种不同癌症类型的不同队列中表现最佳,进一步,研究团队探索了NetBio预测是否可以重现与免疫治疗反应相关的免疫微环境(图5)。结果显示,NetBio的预测成功地再现了免疫微环境。Gide和Liu数据集(黑色素瘤队列)表现出相似的免疫微环境特征,与白细胞比例、CD8 T细胞比例呈高度正相关,与M2巨噬细胞比例呈高度负相关。研究团队还分析了与免疫细胞比例高度相关的NetBio通路,发现FGFR信号通路与CD8 T细胞比例的相关性最高,该通路的表达水平与细胞比例呈负相关。

图5. NetBio再现免疫微环境。来源:Nature Communications
研究团队使用了不同的免疫表型,并将其与趋化和吞噬通路的表达水平进行了比较。经验证,在PD-L1治疗的膀胱癌队列中,趋化和吞噬相关通路与免疫浸润相关,NetBio揭示了与免疫治疗反应相关的免疫微环境通路。同时,将NetBio与ML模型的肿瘤突变负荷(TMB)相结合可以提高PD-L1单抗治疗膀胱癌患者的总体生存预测能力。
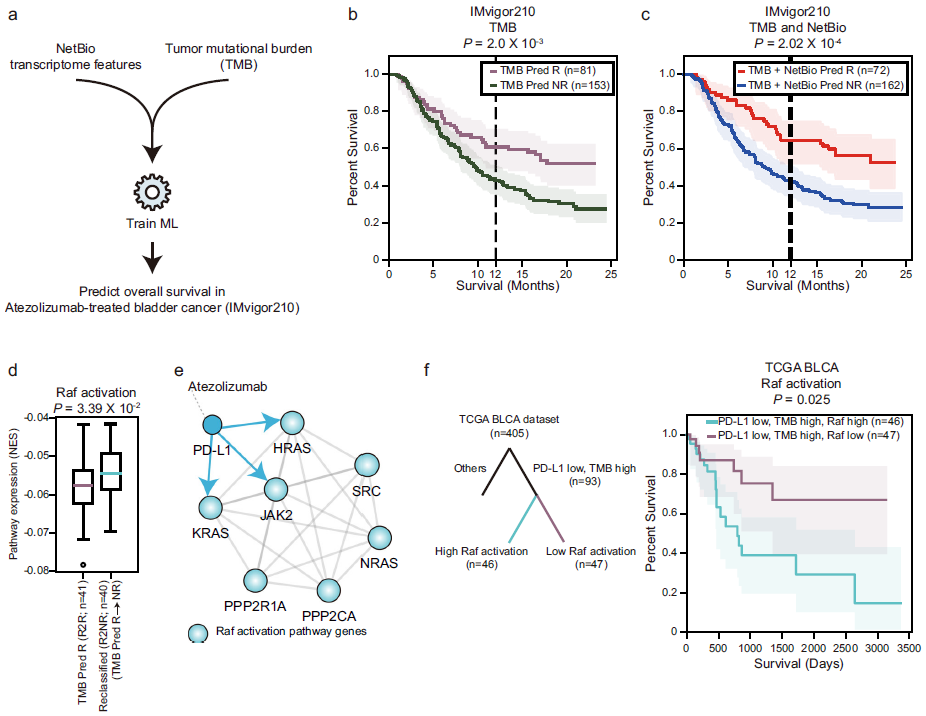
图6. NetBio可优化膀胱癌患者的总体生存预测。来源:Nature Communications
结 语
综上所述,该研究团队测试了基于网络的生物标志物能否对免疫治疗做出稳健的预测。结果显示,基于NetBio的ML表现出一致的优良预测性能,并且优于纯数据驱动方法识别的特征。利用PPI网络的确能够增加对稳健生物标志物的检测。这一研究有助于提前发现对免疫治疗有反应的患者,并制定个性化治疗计划,让更多的患者受益于癌症免疫疗法。
参考文献:
1. JungHo Kong et al, Network-based machine learning approach to predict immunotherapy response in cancer patients, Nature Communications (2022).
2. Havel, J. J., Chowell, D. & Chan, T. A. The evolving landscape of biomarkers for checkpoint inhibitor immunotherapy. Nat. Rev. Cancer 19, 133–150 (2019).
3. Lakatos, E. et al. Evolutionary dynamics of neoantigens in growing tumors. Nat. Genet. 52, 1057–1066 (2020).
·END ·
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#Nat#
113
#预测癌症#
70
#标志物#
65
#COMMUN#
75
#癌症患者#
106
#生物标志#
62
#生物标志#
68
#机器#
86
#生物学#
82