Mol Cell:谢明艺团队揭示了核内小RNA 7SK 通过m6A修饰调控细胞转录水平的分子机制
2023-10-14 iNature iNature 发表于上海
该研究揭示了m6A通过调控7SK snRNA介导的转录延伸复合物(pTEFb)的释放,从而调控了细胞的转录延伸,最终影响了细胞的整体转录水平。
N6-甲基腺嘌呤(m6A)是真核生物信使RNA(mRNA)内部修饰中最丰富的一种,主要由METTL3/METTL14/WTAP/RBM15甲基化酶复合体(writer)介导,而去甲基化酶ALKBH5和FTO(eraser)负责动态、可逆的去甲基化1-7。m6A通过其特异性识别蛋白(reader)参与了RNA代谢的多个方面1,8,9。除了存在于mRNA上,m6A也广泛存在于非编码RNA上。然而,过去十年来对m6A的研究主要集中在修饰丰度较高的mRNA上,探讨了其在转录后调控mRNA剪接、转运、翻译以及稳定性等方面。近期研究表明m6A通过调控与染色质相关的RNA(如启动子RNA、转座子原件的重复RNA、增强子RNA)的稳定性,从而调控了细胞的整体转录水平1,10,11。然而,m6A是否也通过长链非编码RNA影响细胞的转录水平,目前尚不明确。
2023年10月10日,美国佛罗里达大学的谢明艺教授团队在Molecular Cell 杂志在线发表了题为“N6-methyladenosine in 7SK small nuclear RNA underlies RNA polymerase II transcription regulation”,揭示了m6A通过调控7SK snRNA介导的转录延伸复合物(pTEFb)的释放,从而调控了细胞的转录延伸,最终影响了细胞的整体转录水平。

研究人员首先通过MeRIP-qRCR实验系统比较了两组正常对照细胞和四组非小细胞肺癌(NSCLC)中7SK snRNA的修饰丰度,结果显示在非小细胞肺癌中,7SK的m6A修饰丰度明显高于正常对照细胞。在NSCLC A549细胞中,研究人员通过SELECT-qPCR技术系统筛选了7SK上的多个m6A修饰位点。为了进一步研究鉴定出的m6A修饰位点的生物学意义,研究人员通过过表达dCasRx-ALKBH5融合蛋白特异性去除7SK上的m6A修饰(见下图)。
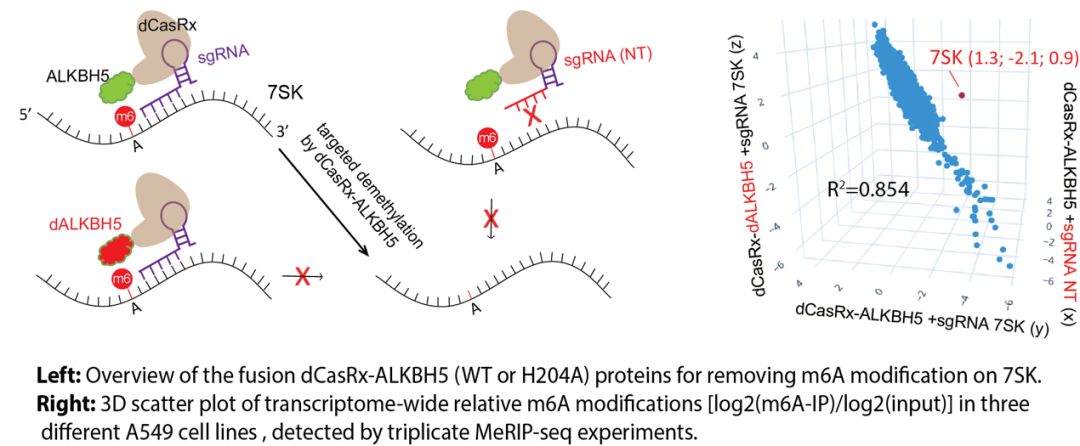
去除7SK m6A修饰显著抑制了RNA Pol II CTD二号位丝氨酸的磷酸化水平及细胞的整体转录水平。研究人员在野生型细胞系及过表达dCas9-ALKBH5融合蛋白的A549细胞系中通过RNA Pol II 及RNA Pol II Ser2P的ChIP-seq实验发现,特异性去除7SK snRNA上的m6A修饰显著增加了Pol II在启动子区域的富集,并显著减少了Pol II在基因体区域的信号。Pol II ChIP-seq结果与Pol II Ser2P ChIP-seq 结果相互印证。此外,研究人员通过Nascent RNA-seq 分析发现,去除7SK snRNA 上的m6A修饰显著增加了未成熟的RNA的丰度(见下图)。
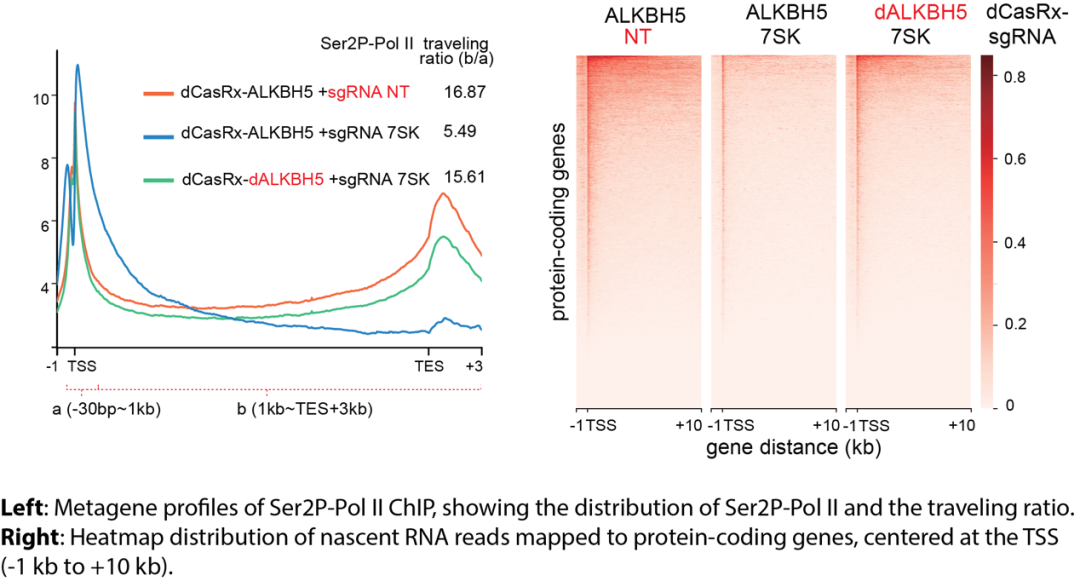
为了进一步研究m6A通过7SK snRNA介导细胞转录延伸的分子机制,研究人员进行了SHAPE-MaP分析。研究结果表明去除7SK snRNA m6A修饰,显著改变了7SK snRNA的结构。且通过Co-IP实验发现7SK snRNA m6A修饰显著抑制了p-TEFb复合体与7SK snRNA的结合。此外,研究人员还发现,特异性去除7SK snRNA m6A修饰,显著抑制了非小细胞肺癌细胞的增殖。
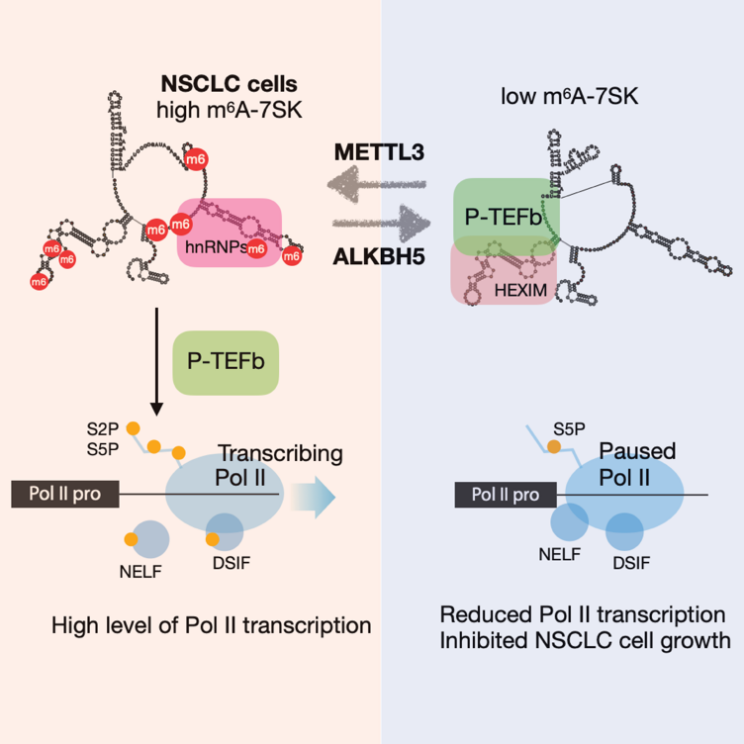
总的来说,该研究通过从7SK snRNA m6A修饰调控细胞转录水平的分子机制入手,结合了ChIP-seq、Nascent RNA-seq、SHAPE-MaP等技术,揭示了m6A通过调控7SK snRNA来调控细胞的转录延伸,从而影响了非小细胞肺癌的增殖机制 (见下图)。
美国佛罗里达大学医学院的博士后王玉芝担任本研究的第一作者,谢明艺教授为通讯作者。此外,感谢实验室其他成员及合作团队在此项目中的鼎力支持。
谢明艺课题组专注于研究癌症和神经疾病模型中非编码RNA介导的基因调控。我们目前正在招募具有生物信息学、分子生物学和癌症生物学背景的博士后研究员,致力于对非编码RNA进行深入研究。欢迎对RNA研究感兴趣的博士生积极报名。请将简历发送至:mingyi.xie@ufl.edu。
参考文献:
1. Liu, J., Dou, X., Chen, C., Chen, C., Liu, C., Xu, M.M., Zhao, S., Shen, B., Gao, Y., and Han, D. (2020). N 6-methyladenosine of chromosome-associated regulatory RNA regulates chromatin state and transcription. Science 367, 580-586.
2. Liu, J., Yue, Y., Han, D., Wang, X., Fu, Y., Zhang, L., Jia, G., Yu, M., Lu, Z., and Deng, X. (2014). A METTL3–METTL14 complex mediates mammalian nuclear RNA N 6-adenosine methylation. Nature chemical biology 10, 93.
3. Ping, X.-L., Sun, B.-F., Wang, L., Xiao, W., Yang, X., Wang, W.-J., Adhikari, S., Shi, Y., Lv, Y., and Chen, Y.-S. (2014). Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase. Cell research 24, 177.
4. Patil, D.P., Chen, C.-K., Pickering, B.F., Chow, A., Jackson, C., Guttman, M., and Jaffrey, S.R. (2016). m 6 A RNA methylation promotes XIST-mediated transcriptional repression. Nature 537, 369.
5. Knuckles, P., Lence, T., Haussmann, I.U., Jacob, D., Kreim, N., Carl, S.H., Masiello, I., Hares, T., Villaseñor, R., and Hess, D. (2018). Zc3h13/Flacc is required for adenosine methylation by bridging the mRNA-binding factor Rbm15/Spenito to the m6A machinery component Wtap/Fl (2) d. Genes & development 32, 415-429.
6. Jia, G., Fu, Y., Zhao, X., Dai, Q., Zheng, G., Yang, Y., Yi, C., Lindahl, T., Pan, T., and Yang, Y.-G. (2011). N6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. Nature chemical biology 7, 885.
7. Zheng, G., Dahl, J.A., Niu, Y., Fedorcsak, P., Huang, C.-M., Li, C.J., Vågbø, C.B., Shi, Y., Wang, W.-L., and Song, S.-H. (2013). ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility. Molecular cell 49, 18-29.
8. Wang, X., Lu, Z., Gomez, A., Hon, G.C., Yue, Y., Han, D., Fu, Y., Parisien, M., Dai, Q., and Jia, G. (2014). N 6-methyladenosine-dependent regulation of messenger RNA stability. Nature 505, 117.
9. Liu, N., Dai, Q., Zheng, G., He, C., Parisien, M., and Pan, T. (2015). N 6-methyladenosine-dependent RNA structural switches regulate RNA–protein interactions. Nature 518, 560.
10. Wei, J., Yu, X., Yang, L., Liu, X., Gao, B., Huang, B., Dou, X., Liu, J., Zou, Z., and Cui, X.-L. (2022). FTO mediates LINE1 m6A demethylation and chromatin regulation in mESCs and mouse development. Science 376, 968-973.
11. Lee, J.-H., Wang, R., Xiong, F., Krakowiak, J., Liao, Z., Nguyen, P.T., Moroz-Omori, E.V., Shao, J., Zhu, X., and Bolt, M.J. (2021). Enhancer RNA m6A methylation facilitates transcriptional condensate formation and gene activation. Molecular Cell 81, 3368-3385. e3369.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








