论文解读|Qi Zhou/Zhou Fu/Tingyuan Lang教授团队新研究建立了一种基于分子特征的卵巢癌生存预测模型
2024-03-06 Genes and Diseases Genes and Diseases 发表于上海
该研究以癌症基因组图谱(TCGA)中的miRNA/mRNA表达数据和临床信息为基础,通过严格的数据预处理和特征选择方法,利用深度神经网络(DNN)开发了一种基于分子特征的卵巢癌生存预测模型。

卵巢癌是一种致死率极高的妇科癌症,准确的生存预测对个性化治疗大有裨益。重庆大学肿瘤医院Qi Zhou、Tingyuan Lang教授团队联合重庆医科大学Zhou Fu教授团队在本刊发表了题为“Development of a molecular feature-based survival prediction model of ovarian cancer using the deep neural network”的研究快讯,该研究以癌症基因组图谱(TCGA)中的miRNA/mRNA表达数据和临床信息为基础,通过严格的数据预处理和特征选择方法,利用深度神经网络(DNN)开发了一种基于分子特征的卵巢癌生存预测模型。主要结果有:
1. 特征提取与生存分析
研究利用Cox比例风险模型分析和Kaplan-Meier算法从TCGA卵巢癌数据中提取与生存相关的特征,识别出与生存相关的172个致癌基因和155个抑癌基因,再根据3年/5年总生存率将患者分为三组具有不同生存结果的类别(图1A、B)。
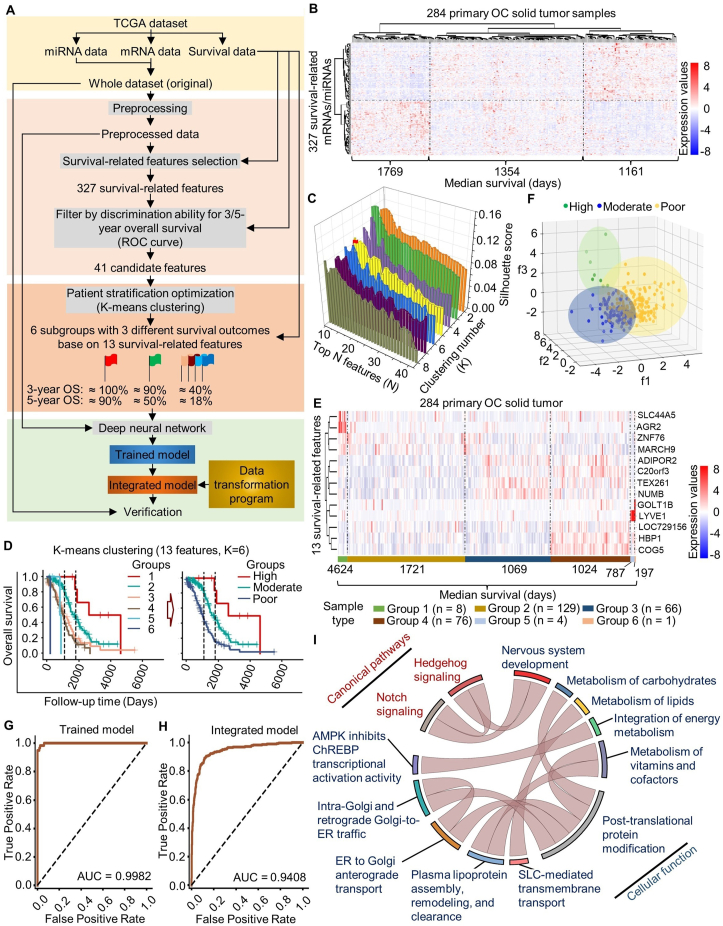
图1 使用深度神经网络的基于分子特征的卵巢癌生存预测模型
2. 最佳聚类与特征组合
作者团队使用K-means聚类算法对患者的生存相关特征进行组合,确定了用于生存预测的mRNA/miRNA特征的最佳组合。结果表明将患者通过13个特征分为6组获得了最好的预测能力。这些群体代表了三种不同的生存结果:高、中、低,中位生存时间存在显著差异,突显出所选特征的有效分组潜力 (图1C–F)。
3. 深度神经网络模型的开发
随后,作者团队用 DNN 开发生存预测模型。将数据8:2分为训练集与测试集,并采用五折交叉验证进行超参数优化,训练模型的平均AUC值为0.9982(图1G)。此外,作者团队还开发了一个集成模型,该模型采用数据转换程序,在真实样本测试中表现出高性能(AUC = 0.9408,图1H),证明了其在预测卵巢癌患者生存结果方面的稳定性和准确性。最后,作者团队对预测模型所使用的13个生存相关基因进行了通路分析,进一步了解生存分析模型辨别能力的分子基础(图1I)。
总之,该预测模型具有高准确性和高效的优点,标志着卵巢癌个性化医学的重大进步,其利用复杂分子数据进行生存预测的能力为临床肿瘤学中的治疗规划和预后评估开辟了新途径。
文章来源
免费全文下载链接:
https://www.sciencedirect.com/science/article/pii/S235230422200280X
引用这篇文章:
Lang T, Yang M, Xia Y, et al. Development of a molecular feature-based survival prediction model of ovarian cancer using the deep neural network. Genes Dis. 2023;10(4):1190-1193.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#卵巢癌# #癌症基因组图谱# #预测模型#
0